روش های مختلف بررسی بیان ژن
تفاوت در تظاهر ژن در تمامی مراحل رشد موجود از تولد تا مرگ اتفاق می افتد. شناسایی ژن های تظاهر یافته، در شناخت عمل ژن و نیز در شناخت سازوکارهای مولکولی یک سیستم بیولوژیک به ما کمک خواهد کرد. در دو دهه پیش مطالعه تظاهر ژن براساس روش های تجزیه یک ژن مانند روش های لکه گذاری نورتن و روش ASO بوده است. در ادامه با ما همراه باشید تا با روش های مختلف بررسی بیان ژن آشنا شویم.
معرفی روش های بررسی بیان ژن
امروزه از روش هایی استفاده می شود که می توانند ژن ها را با تظاهر متفاوت در بین نمونه های آزمایشی شناسایی کنند. مهم ترین این روش ها عبارت اند از:
- دورگ گیری کاهنده (Subtractive hybridization)
- PCR آغازگر اختیاری RNA
- RT-PCR افتراقی
- cDNA-AFLP
- ردیف تظاهر یافته برچسب دار
- ردیف یابی نقاط نشانمند از توالی تظاهر یافته
- تجزیه ترتیبی تظاهر ژن
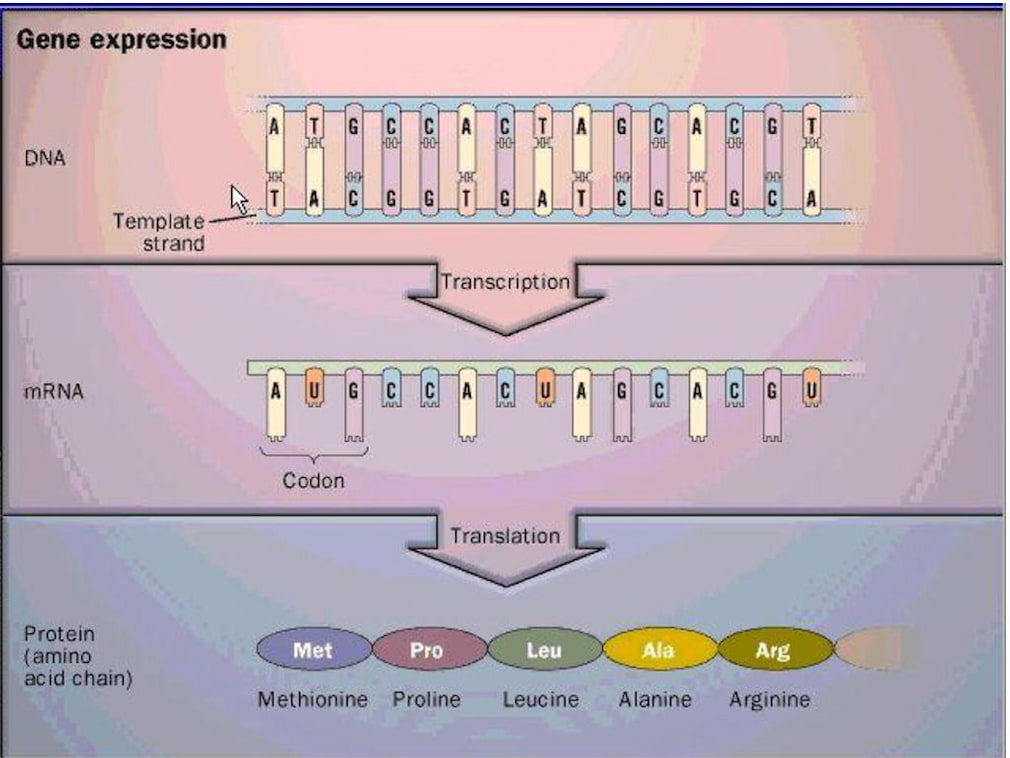
معرفی روش های بررسی بیان ژن
۱- روش دورگ گیری کاهنده
این روش نخستین بار توسط سرگنت و دیوید (۱۹۸۳) به منظور ایجاد کتابخانه cDNA را و پس از آن توسط دیویس و همکاران (۱۹۸۴) به منظور ایجاد کاوشگر از ژن های با تظاهر متمایز استفاده شد. در روش دورگ گیری کاهنده، ژن های با تظاهر متمایز از طریق دورگ گیری cDNA یک نمونه (تستر) با mRNA نمونه دوم (درایور) جدا می شوند. رونوشت های تظاهر یافته در نمونه اول و دوم، دورگ cDNA با mRNA ایجاد می کنند. در حالی که cDNA خاص تستر به صورت تک رشته باقی خواهد ماند. مولکول های تک رشته ای و دو رشته ای با استفاده از کروماتوگرافی از همدیگر جدا خواهند شد.
مولکول های تک رشته ای cDNA جدا شده، ژن هایی را نشان می دهند که فقط در نمونه تستر تظاهر یافته اند. این cDNA ها را می توان همسانه کرد و از آنها به طور مستقیم به عنوان کاوشگر هایی برای غربال کتابخانه استفاده نمود. به منظور افزایش کارایی روش دورگ گیری کاهنده اصلاحاتی در این روش ایجاد شده است. برای جداسازی بهتر مولکول های تک رشته ای از مولکول های دو رشته ای استفاده از cDNA های با برچسب های بیوتین یا الیکو (dT) مطرح شد. همچنین به منظور کاهش مقدار RNA اولیه مورد نیاز و بهبود کارایی همسانه سازی، تکثیر cDNA انتخاب شده از طریق PCR توسط هارا (۱۹۹۱) مدنظر قرار گرفت.
۲- پی.سی.آر آغازگر اختیاری آر.ان.ا (RAP-PCR)
PCR آغازگر اختیاری RNA در حقیقت یک روش استفاده از AP-PCR بر روی RNA است و برای بررسی تغییرات تظاهر ژن های مختلف استفاده می شود. همچنانکه پیشتر توضیح داده شد، AP-PCR یکی از روش های مفید برای شناسایی انگشت نگاری در سطح DNA موجود است. در این روش از آغازگرهای اختیاریی استفاده می شود که حتی در حالت عدم جفت شدن کامل با DNA الگو می توانند DNA را سنتز کنند.
از روش AP-PCR برای انگشت نگاری آران را به منظور شناسایی تفاوت در بیان ژن نیز می توان استفاده کرد، که به آن RAP- PCR گفته می شود. در این روش از آغازگر قلاب شده در انتهای ‘۵ مانند dT) CA oligo) به منظور رونوشت برداری معکوس و از یک آغازگر اختیاری برای اتصال به دومین رشته cDNA استفاده می شود. شدت بانددهی RAP- PCR در بین نمونه ها، با مقدار فراوانی RNA و در نتیجه با تفاوت در تظاهر ژن ارتباط دارد.
در انتخاب آغازگرهای RAP- PCR (و همچنین AP-PCR) باید به نکات زیر توجه کرد:
- آغازگر باید فاقد ساختار ثانویه پایدار باشد؛
- ردیف آغازگر باید طوری انتخاب شود که انتهای ‘۳ آن مکمل ردیف دیگر در آغازگر نباشد؛
- آغازگرهای ۱۰ تا ۲۰ نوکلئوتیدی بسیار مناسب تر.
آغازگرهای ۲۰ نوکلئوتیدی این مزیت را نسبت به آغازگرهای کوچک تر دارند که اگر در همسانه سازی و ردیف یابی استفاده شوند، اطلاعات ردیف بیشتری دارند. اما مزیت آغازگرهای ۱۰ نوکلئوتیدی ارزان تر بودن آنهاست. اگر هدف بدست آوردن قطعه های زیاد باشد باید:
- در غربال اولیه از آغازگرهای ۱۰ نوکلئوتیدی که با هولو آنزیم آمپلی تگ قطعه های زیادی ایجاد می کند استفاده شود.
- از آغازگرهای ۱۰ تایی با قطعه استافل استفاده گردد.
- در صورت استفاده از آغازگرهای بسیار کوچک غلظت آنها بیشتر شود.
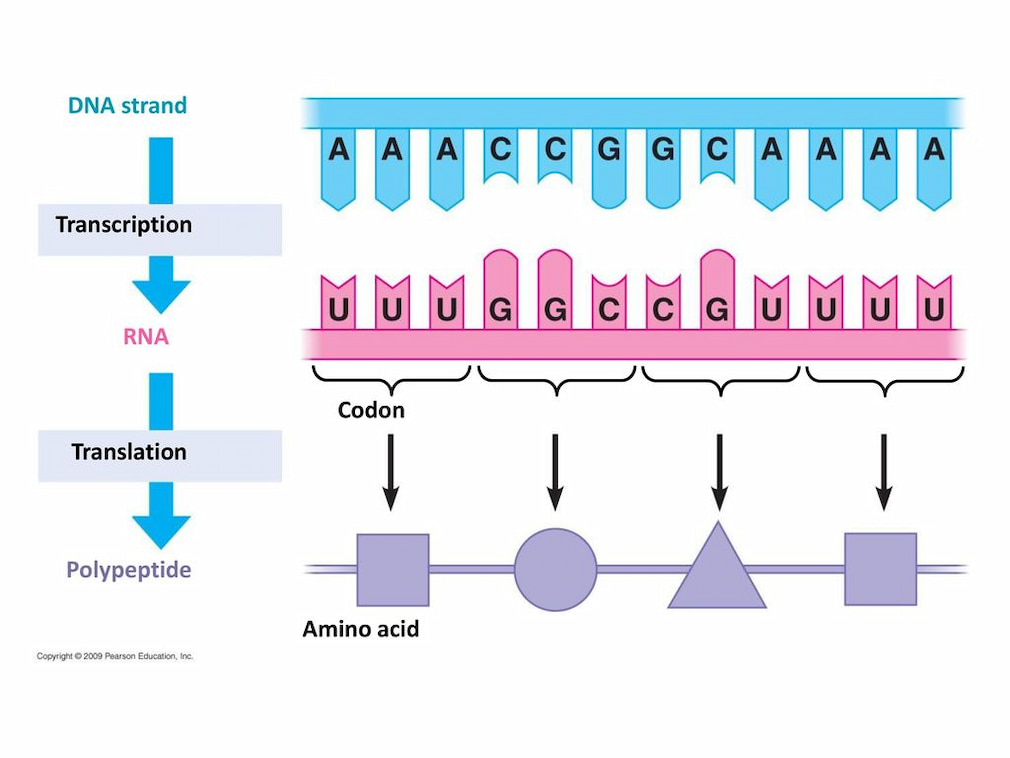
بررسی بیان ژن
۳- RT-PCR افتراقی (DDRT)
RT-PCR افتراقی که در سال ۱۹۹۲ توسط لیانگ و پادری مطرح گردید، روش مناسبی برای بررسی بیان ژن هاست. در حقیقت این روش یک روش انگشت نگاری برای شناسایی و مقایسه mRNA های بیان شده در طول مراحل مختلف سلول است. برای مثال روش بسیار مفیدی برای شناسایی ژن هایی است که در بافت های خاص (مانند بافت های سرطانی) در واکنش به دارو یا آلودگی ویروسی تظاهر می یابند. همچنین روش مفیدی برای شناسایی mRNA هایی است که در شرایط استرس و شوک ایجاد می شوند.
-
معایب روش RT-PCR افتراقی
با وجود مزایای زیاد روش RT-PCR افتراقی، این روش معایبی نیز دارد. که مهمترین آنها ایجاد اشتباه مثبت در مرحله PCR است. همچنین از آنجا که قطعه cDNA تکثیر شده عمدتا مطابق با ناحیه انتهای mRNA است، بنابراین شناسایی ژن هایی که تظاهر متفاوت دارند محدود خواهد شد. از این رو تغییراتی در این روش به وجود آمده است که از آن میان، می توان افزایش طول آغازگر مورد استفاده (به جای آغازگرهای ۱۰ نوکلئوتیدی) برای افزایش اختصاصی بودن و تکرارپذیری و همچنین ایجاد تغییر در آغازگرها برای سهولت مراحل همسانه سازی و تجزیه قطعه ها را نام برد. به طوری که PCR در دو مرحله انجام می گیرد، در مرحله نخست که شامل چندین چرخه با سختی کم است، عدم جفت شدن کامل بین آغازگر اختیاری و الگوی cDNA را به وفور اتفاق می افتد.
در مرحله دوم که شامل تعداد بیشتری چرخه PCR با سختی زیاد است، تطابق کامل با آغازگرهای قلاب شده اتفاق می افتد. همچنین از آنجا که ردیف یابی انجام گرفته روی محصولات PCR بیشتر مربوط به ناحیه ترجمه نشده است، با افزایش طول آغازگر محصول PCR بزرگ تری بدست خواهد آمد. به این ترتیب، در نهایت ردیف های طویل تری به دست خواهند آمد که برای بررسی همولوژی از طریق بانک های اطلاعاتی مفید خواهند بود.
۴- روش cDNA-AFLP از روش های بررسی بیان ژن
این روش که توسط بچم و همکاران (۱۹۹۶) معرفی شد، یک روش تکرارپذیر مناسب برای آشکارسازی رونوشت های با تظاهر متمایز است و بدلیل تکرارپذیری و حساسیت و از طرفی به علت تطابق خوب با روش لکه گذاری نورتن، به وفور مورد استفاده قرار می گیرد. این روش شامل مراحل زیر است:
- ساختن cDNA با استفاده از الیگونوکلئوتید پلی (dT)؛
- ایجاد الگوی اولیه از طریق برش با دو آنزیم محدود کننده و اتصال قلاب ها به انتهای آنهاء
- تکثیر قبل از مرحله تکثیر اصلی به کمک آغازگرهایی که بر اساس نواحی قلاب (لنگر) شده طراحی شده اند؛
- تکثیر اصلی با آغازگرهایی که یک یا دو باز اختصاصی دارند. یکی از این آغازگرها نشاندار است تا محصول نهایی قابل رویت باشد.
۵- ردیف تظاهر یافته برچسب دار یا EST
عدم دسترسی به کاوشگرها یا همسانه ها سبب محدود شدن استفاده از نشانگرهای RFLP می شود. از این رو امروزه جایگاه های ژنی RFLP به STS یا EST تبدیل می شود. EST ردیف های ترجمه شده یا ترجمه نشده را نشان می دهد، در صورتیکه EST ردیفی از یک همسانه cDNA را نشان می دهد که با یک مولکول mRNA یا قسمتی از آن مطابقت دارد. از EST های با طول ۵۰۰-۱۵۰ جفت باز برای بررسی تشابه و همچنین نقشه یابی استفاده می شود.
در حقیقت می توان از mRNAهای ایجاد شده در بافت های مختلف و در شرایط گوناگون cDNA هایی را ایجاد کرد و سپس برخی از این cDNAها را ردیف یابی نمود که البته بیشتر ردیف یابی انتهای ‘۳ یا ‘۵ همسانه های cDNA صورت می گیرد که EST نامیده می شود. به عبارت دیگر، EST ردیف های DNA انتهای ‘۳ یا ‘۵ همسانه های cDNA هستند که اغلب به طور تصادفی و یا بر اساس غربال کردن همسانه های خاص از یک کتابخانه cDNA را انتخاب می شوند. بنابراین هر cDNA، دو EST می تواند داشته باشد، یکی مطابق با انتهای ‘۳ و دیگری مطابق با انتهای ‘۵ آن cDNA، اما در بیشتر موارد ناحیه بین دو EST ناشناخته است.
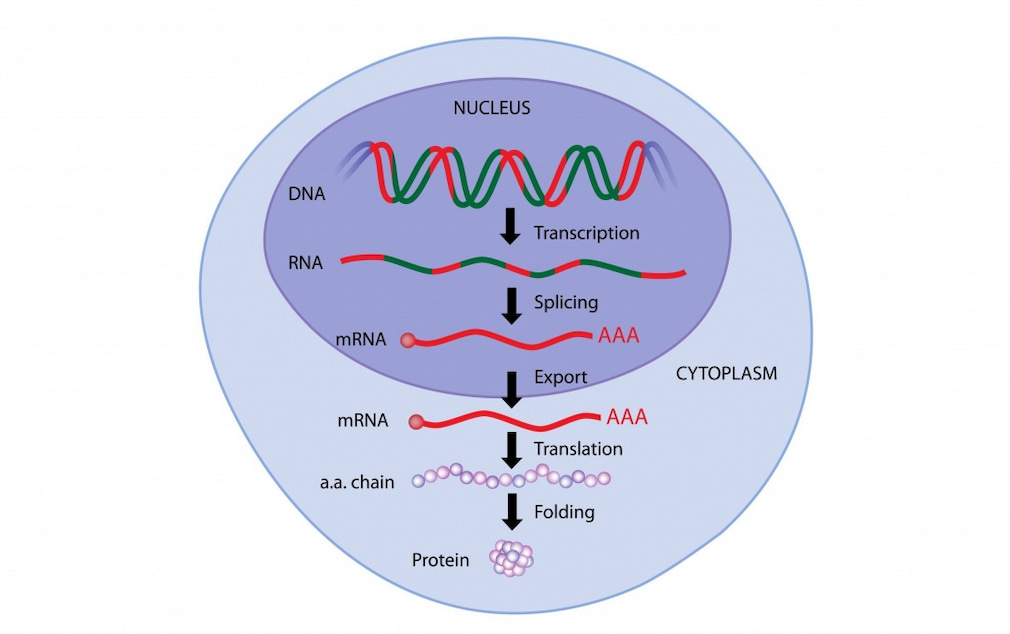
بیان ژن
۶- ردیف یابی نقاط نشانمند از توالی تظاهریافته
اساس این روش بر این اصل است که سطح یک mRNA را در یک بافت خاص از طریق فراوانی EST منطبق با آن در یک کتابخانه سی.دی.ان را منعکس می شود. همچنان که بیان شد، ردیف تظاهر یافته برچسب دار یا EST از طریق ردیف یابی انتهای ‘۳ یا ‘۵ همسانه های cDNA بدست می آید. تفاوت در تظاهر ژن از طریق شمارش دفعات ظهور یک ردیف خاص در کتابخانه EST ژن های منابع مختلف، مشخص می شود.
EST های بدست آمده از کتابخانه های cDNA نرمال شده، با همدیگر مقایسه می شوند و براساس آن رونوشت هایی که در یک کتابخانه تظاهر می یابند، ولی در کتابخانه دیگر وجود ندارند، شناسایی می شود. با وجود این، فراوانی نسبی ژن ها در یک کتابخانه، فقط به کمک EST های به دست آمده از کتابخانه های cDNA نرمال نشده قابل تعیین خواهد بود.
۷- تجزیه ترتیبی تظاهر ژن (SAGE) از روش های بررسی بیان ژن
این روش که توسط ولکولسکو و همکارن (۱۹۹۵) معرفی شد بر این اساس استوار است که ردیف های برچسب دار کوچک برای شناسایی رونوشت ژن کفایت میکنند. یک برچسب تجزیه ترتیبی بیان ژن از طریق ردیف یابی یک ناحیه معین و کوچکی از یک رونوشت mRNA را به دست می آید. به عبارت دیگر یک برچسب تجزیه ترتیبی بیان ژن را تعیین کرد. از آنجا که از نظر تئوری هر برچسب خاص تجزیه ترتیبی تظاهر ژن، یک رونوشت خاص mRNA را نشان میدهد، بنابراین مجموع ژن های تظاهر یافته در نمونه mRNA اولیه به وسیله ردیف های برچسب ایجاد می گردند.
در نهایت تفاوت در بیان ژن نمونه های مختلف از طریق مقایسه فراوانی نسبی برچسب های ویژه تجزیه ترتیبی بیان ژن در کتابخانه های مختلف قابل شناسایی خواهد بود. ژن ها یا EST هایی که به وسیله برچسب های تجزیه ترتیبی بیان ژن نشان داده می شوند از طریق جست و جو در بانک های اطلاعاتی قابل شناسایی اند.
-
مزایای تجزیه ترتیبی تظاهر ژن (SAGE)
مهمترین مزیت روش تجزیه ترتیبی بیان ژن نسبت به روش های دورگ گیری کاهنده و RT-PCR افتراقی، این است که داده های تجزیه ترتیبی تظاهر ژن کمی و تجمعی هستند. در حقیقت تعیین فراوانی دقیق تمامی ژن های تظاهر یافته در یک سلول و یا بافت از طریق تجزیه ترتیبی بیان ژن موجب تکمیل ردیف یابی می گردد. به طوری که داده های به دست آمده، قابل مقایسه با اطلاعات موجود یا اطلاعات آینده خواهند بود.
مزیت یا ویژگی دیگر روش تجزیه ترتیبی بیان ژن این است که به کمک آن می توان اطلاعات با داده های EST به دست آمده از کتابخانه های کاهنده یا نرمال شده را تکمیل کرد. در حقیقت ردیف های EST، ردیف و اطلاعات حاشیه ای را تعیین می کند، در حالی که روش تجزیه ترتیبی بیان ژن کمیت داده ها و فراوانی ردیف ها را مشخص می کند. مزیت دیگر روش تجزیه ترتیبی بیان ژن این است که مقدار داده های مربوط به بیان ژن که از طریق ردیف یابی یک کتابخانه به دست می آید، تقریبا ۲۰ مرتبه بیشتر از ردیف یابی EST است.
منبع: کتاب نشانگرهای مولکولی، انتشارات دانشگاه تهران










دیدگاهتان را بنویسید